点击蓝字
关注我们
CRISPR/Cas是基因编辑领域中的一项重要技术,近年来随着测序技术发展逐渐成熟和成本的不断下降,其在治疗性基因编辑方面的临床研究日益增加,已经受到越来越多的关注。但是CRISPR会对目标DNA进行双链切割产生双链断裂(double-strand breaks,DSB),DSB会激活细胞自身的双链断裂修复机制,这个修复机制往往会引入各种意想不到的突变,例如:基因序列发生插入或缺失、倒位和拷贝数变异等,这些意外突变往往会导致癌症风险。此外,CRISPR还有较高的脱靶编辑的比例,也会对非目标的DNA区域进行编辑。为了解决这些问题,David Liu的团队开发了一种名为prime editing(PE)的新技术。来自中国科学院的Zhangrao Huang在最近的一篇文章中阐述了PE的发展和应用,本文借此简要介绍该基因编辑系统,并整理了一些PE在多物种的应用情况和目前为提高PE系统编辑效率和特异性的优化策略,为大家进行相关研究提供一些参考。


1
PE系统的简介
PE是一种精确的基因组编辑技术,它依赖于“搜索”和“替换”目标序列。和CRISPR/Cas类似,PE是由一个三元复合物组成的,主要包括nCas9(H840A)、莫洛尼鼠白血病病毒逆转录酶(M-MLV-RT)和pegRNA组成。pegRNA的5’端是有一段特异的sgRNA序列用于引导三元复合与基因组靶位点结合,3’端有一段PBS序列(primer binding site,用于与互补链集合,并起到引物的作用)和RRT序列(reverse transcription template,具有所需编辑序列的逆转录模板)。在PE系统中,pegRNA的5’端将三元复合引导到基因组靶位点与其一个单链特异结合,nCas9对其切割产生一个缺口,3’端的PBS与互补链结合起到引物的作用,通过逆转录酶将RRT上的目的序列逆转录入缺口位置,最终修复得到目的序列(见图1)。
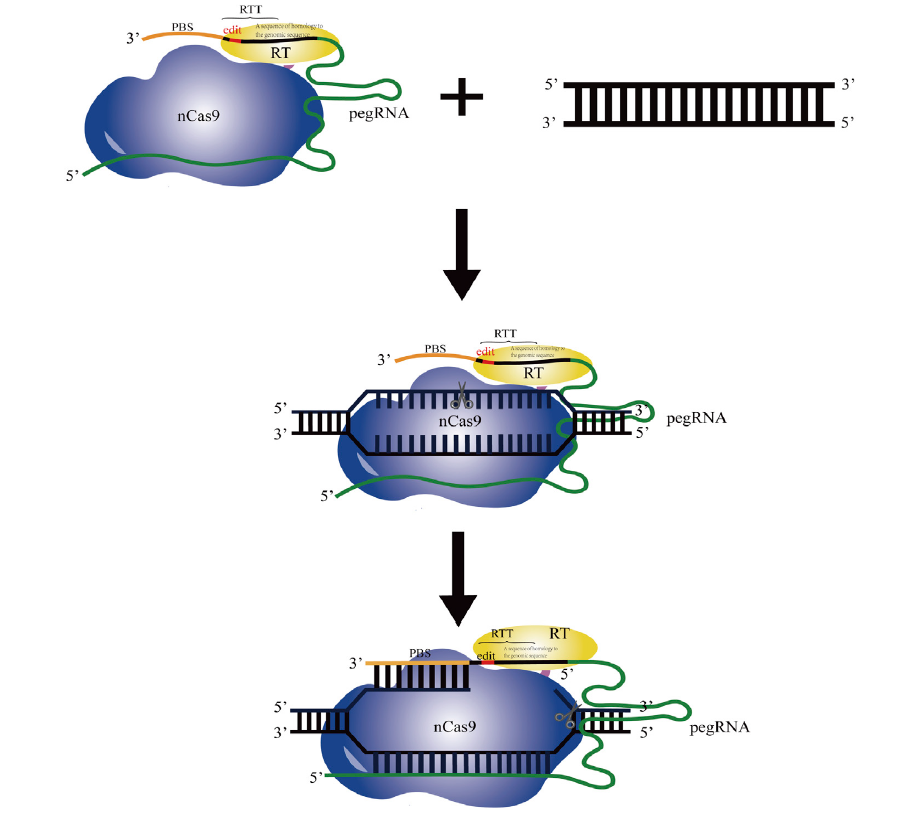
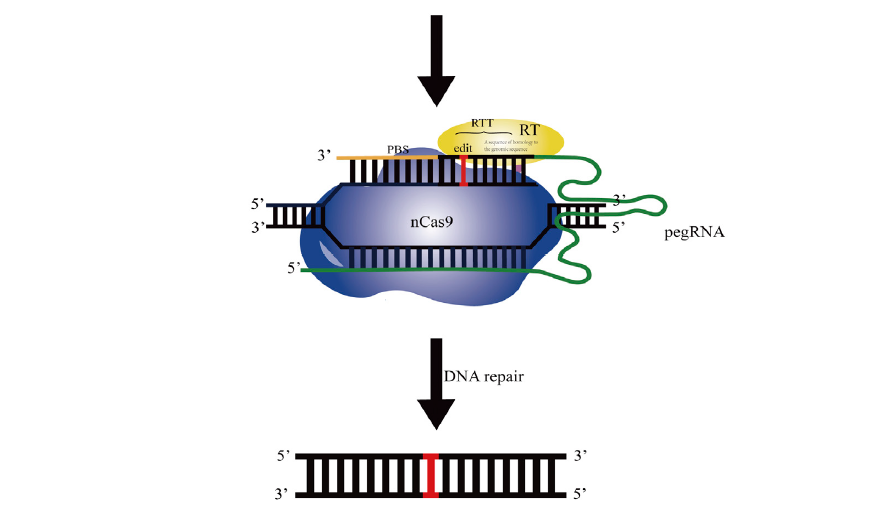
图1

2
PE系统的应用
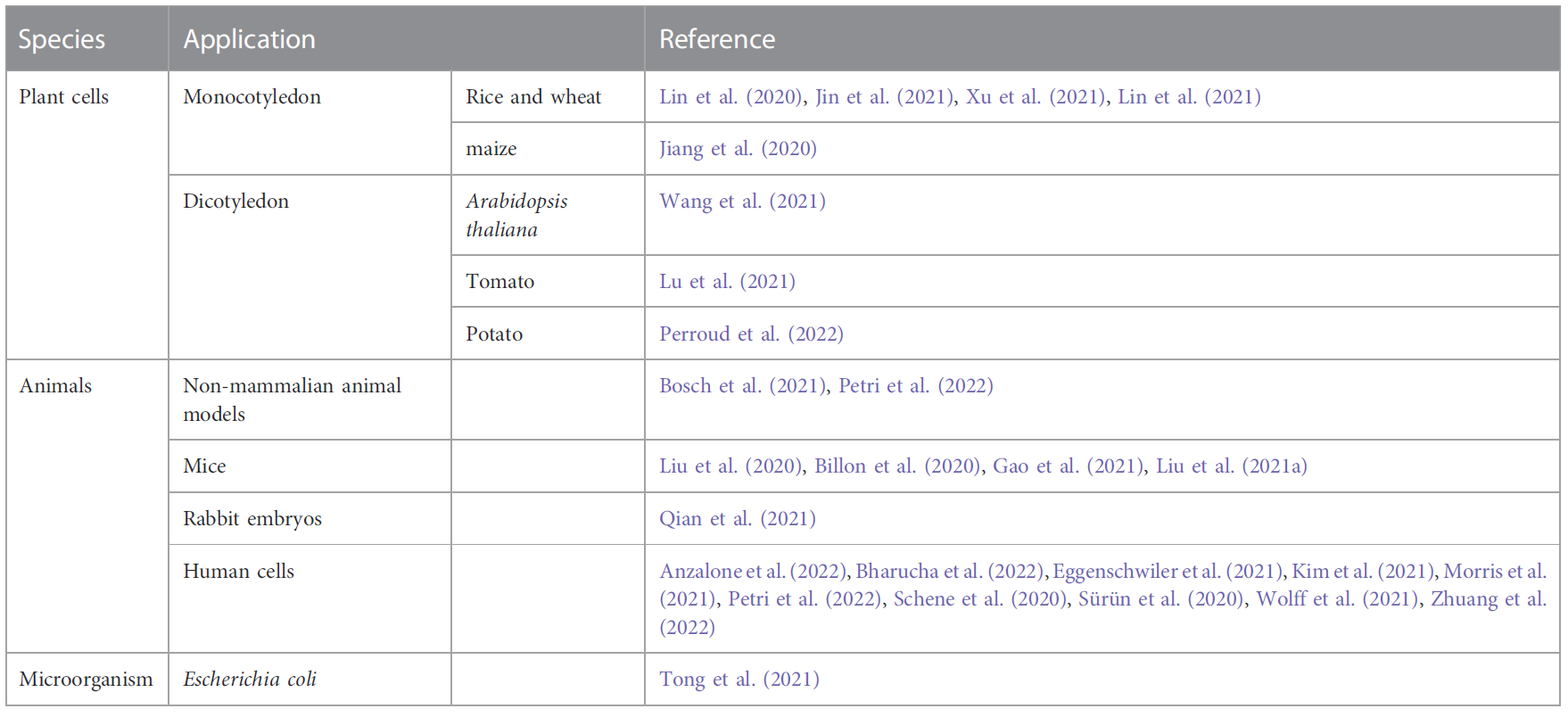
PE技术自问世以来,在多个物种中展现出广泛的应用潜力,更多地被应用于活细胞的生命工程中(见表1)。它适用于12种常见的基因编辑,主要包括点突变、小的片段敲除和小的片段插入,其中插入的基因编辑最多可以到达44bp,敲除的基因编辑最多可以敲除80bp。PE首先应用于哺乳动物细胞,在最初的PE系统中发现,它可以纠正高达89%的与人类疾病相关的已知基因变异[1]。除了在哺乳动物细胞的应用,PE的衍生技术迅速发展和应用于植物,使得原来受限于CPRISPR/Cas技术同源重组效率低和供体传导效率低的问题有效提高了编辑效率[2]。微生物不仅被用作研究生命科学的工具,而且与人类健康、工业和农业生产密切相关。最近,PE也被应用于模型微生物大肠杆菌[3]。
表1 PE技术在生命工程中的应用

3
PE系统在大肠杆菌中的应用
由于大多数原核生物缺乏非同源端连接(NHEJ)系统,基于CRISPR/Cas9系统的早期基因组编辑依赖于同源DNA供体(donor)的存在,这增加了大肠杆菌基因组编辑的难度。因此,基于PE系统的大肠杆菌等原核微生物基因组编辑试剂盒被研发出来。它主要是由pCDF-GFPplus、 pPEgRNA和 pCRISPR-PE三个质粒组成(见图2),pCDF-GFPplus作为报告质粒来评价编辑效率[3]。此外,对PE系统的编辑条件进行了优化,包括诱导条件、PBS的最优长度和RTT的最优值等,并通过检测乳糖代谢通路和D-半乳糖醇代谢通路的突变来评估优化后PE系统的编辑能力。在最佳条件下,单碱基敲除效率高达40%。此外,成功在大肠杆菌中敲除了长达97个碱基的DNA序列,并插入了长达33bp的序列。但是,在大肠杆菌中PE的编辑效率仍然很低,尚有优化的空间。
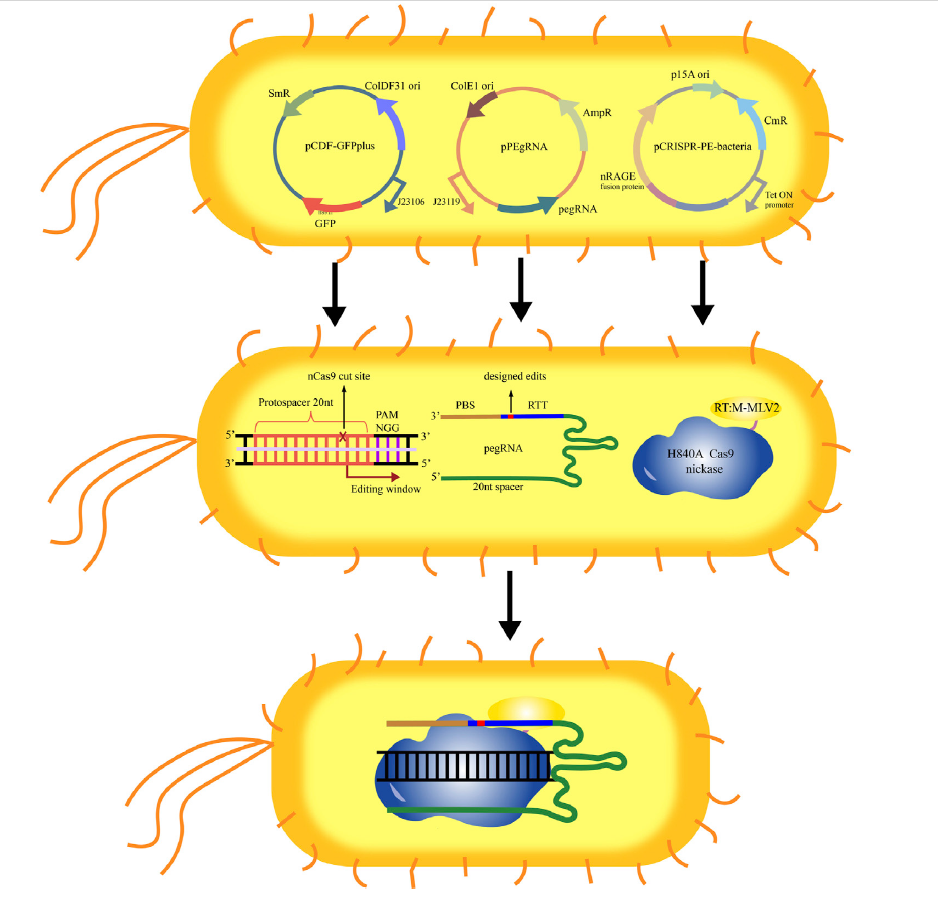
图2

4
PE系统的优化
虽然PE已经应用于动植物以及微生物中,但它的编辑效率和精准性仍然需要进一步优化。当前优化的方向主要就是针对pegRNA、nCas9、逆转录酶等几个主要元件。

4.1 pegRNA的优化
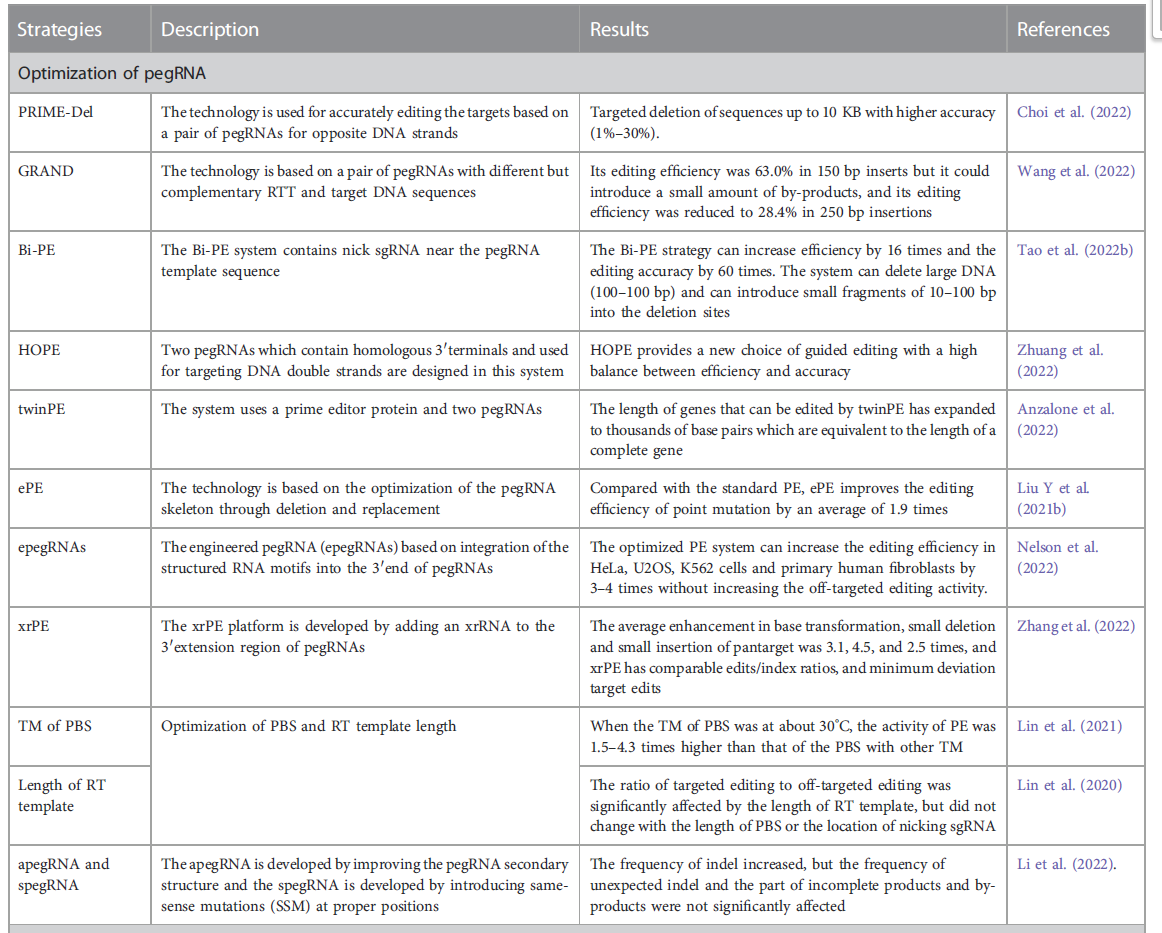
1)在PRIME Del进行PE的时候会加入两个pegRNA,这两个的pegRNA的5’端的sgRNA序列是反向互补的(见图3),会分别识别一个DNA位点的互补的两条DNA单链并引导nCas9分别产生一个缺口。这样可以显著提高PE系统靶向敲除的精准性和灵活性,并且相较于CRISPR/Cas9系统在敲除10kb以上的序列时有更高的效率,效率可以达1%至30%。
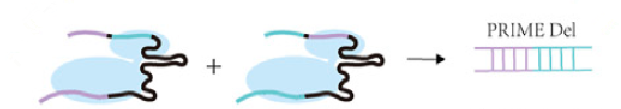
图3
2)在GRAND editing中会加入一对RTT上游序列是反向互补的pegRNA,由于转录进入的DNA和敲除的序列是在这两个间隙间(见图4),这样可以避免基因组中原始序列与新的转录序列之间的竞争提高编辑效率。该系统对150bp DNA片段的插入效率为63.0%,250bp DNA片段的插入效率为28.4%,但是在400bp以上的插入效率是很低的。
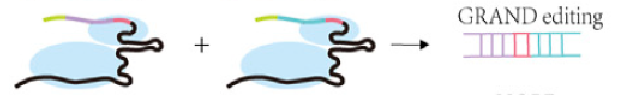
图4
3)在HOPE中会加入一对在3’端同源介导的启动子的pegRNA,它俩的5’端分别是目的位点DNA的正义序列和反义序列[4]。它提供了一种新的编辑方案,并且有较低的脱靶率(见图5)。
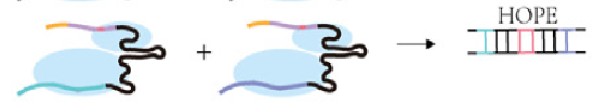
图5
4)在接近pegRNA模板序列位置定位缺口sgRNA有助于靶向敲除大片段的[5],为此Bi-PE被研发出来了。Bi-PE有两个pegRNA,一个会带着间隔序列和模板序列来引导产生一个单链断裂并进行随后延伸,另一个则会对互补链产生一个缺口(见图6),这使得编辑效率提高了16倍,编辑产品的精度提高了60倍。并且Bi-PE可有效地敲除数百至数千碱基对的片段,并且可以同时在缺失的地方引入10bp到100bp片段。
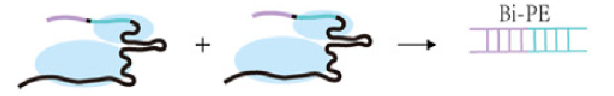
图6
5)但是在100bp以上的碱基对的PE基因编辑效率却很低,因此专家又研发了twinPE。twinPE是两个pegRNA共同使用一个prime的编辑蛋白,它俩分别会对同一个编辑位点上下各自产生一个缺口(见图7),这样可以避免产生DSB,并且将编辑长度延长到数千个碱基对,由于很多基因都只有数千bp,因此对全基因的敲除等类型的基因编辑具有重要意义。此外,当twinPE系统与位点特异性重组酶结合时,成功编辑了与人类细胞中亨特综合征相关的近40 kb倒置的DNA片段。

图7
6)pegRNA的稳定性在很大程度影响着编辑效率,稳定性越高编辑效率越高。而pegRNA主要是从它的3’端开始被降解的,在3’端增加三级结构的序列可以有效保护pegRNA不容易被降解(见图8),因此epegRNA就此被研发出来。epegRNA的编辑效率可提升原有编辑效率的1.1倍至3.3倍。
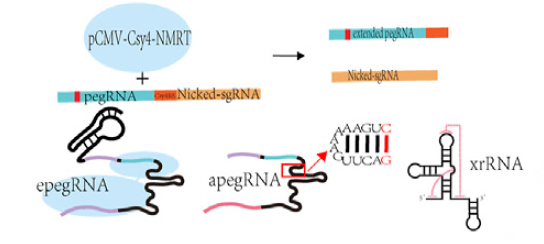
图8

4.2 PE系统的其他优化
1)PBS的长度(6-16nt)、RT模板的长度(7-23nt)和切割sgRNA的位置对编辑效率有很大的影响[6]。当使用不同的PBS长度或不同的sgRNA切割位置时,目标编辑与脱靶编辑的比例没有变化,但它受到RTT长度的显著影响(见图9B)。
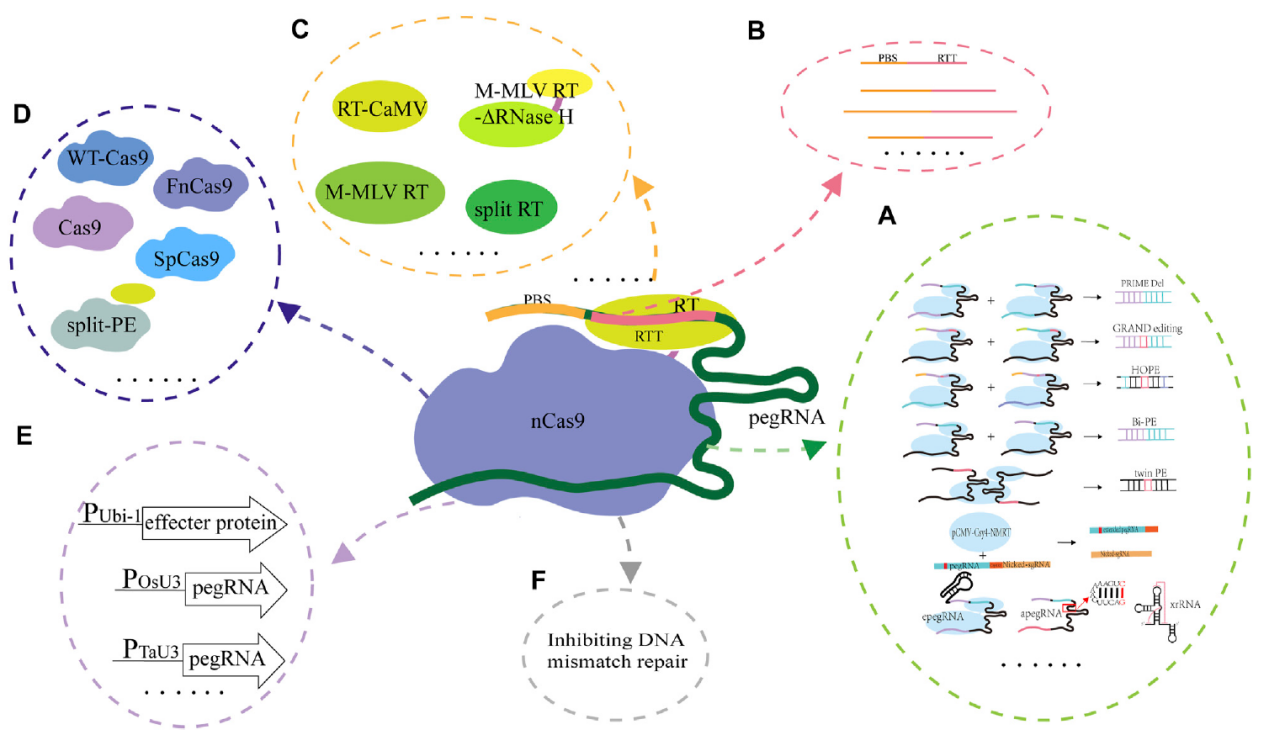
图9
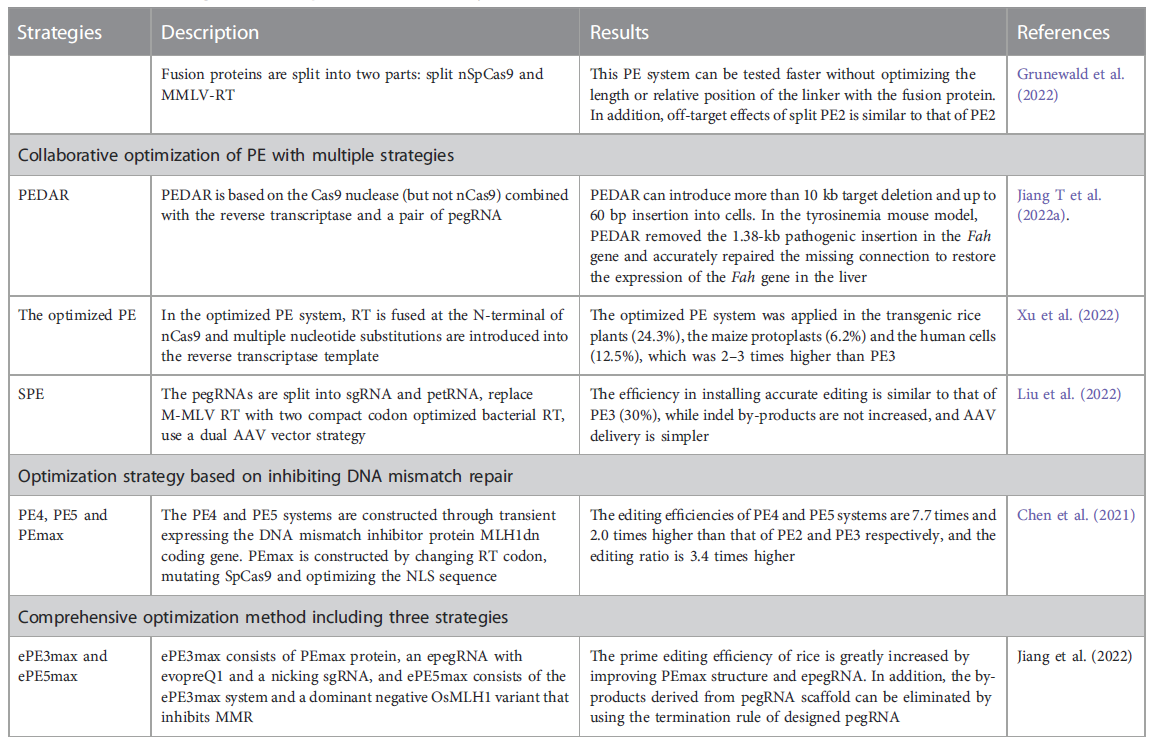
2)逆转录酶的优化:在ePPE系统中,删除了M-MLV RT中的RNase H结构域并添加了病毒核衣壳蛋白(NC);在PPE系统中,使用来自花椰菜花叶病毒的CaMV-RT和来自大肠杆菌BL21的反转录酶衍生的RT(RT retron)替换了工程化的M-MLV-RT;在Spilt-PE中,将融合效应蛋白拆分为两部分,包括拆分的nSpCas9和MMLV-RT(见图9C)。
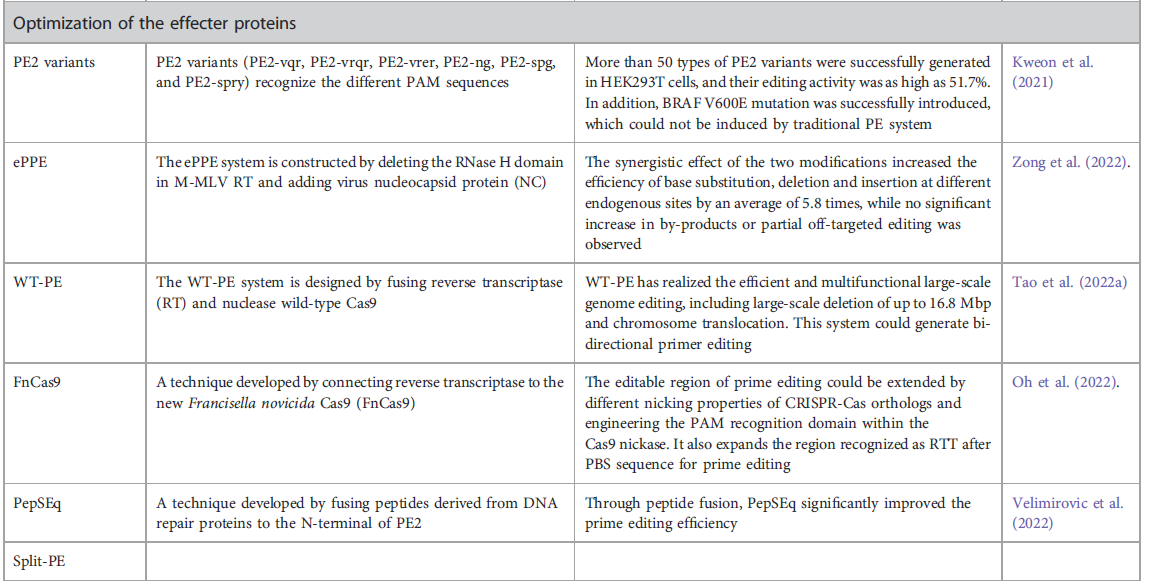
3) 扩大Cas9的编辑范围的优化:PE2的变体(PE2-vqr、PE2-vrqr、PE2-vrer、PE2-ng、PE2-spg和PE2-spry)可以识别不同的PAM序列(见图9D)。
4)效应蛋白基因和pegRNA表达启动子的优化:在PPE中,使用玉米的泛素-1基因(Ubi-1)启动子来驱动经过水稻密码子偏好性优化的效应蛋白编码基因的表达;使用水稻的osu3启动子和小麦的TaU6启动子来驱动pegRNA和切割sgRNA的转录(见图9E)。
5) 通过抑制DNA错配修复来优化PE系统:生物体种中DNA一条单链突变时,互补链容易通过DNA的错配修复机制(mismacth repair,MMR)修复成相同的突变或者是野生型(见图10红线框部分)。因此PE系统中为了抑制MMR,得到目的基因编辑须进行相关优化。通过使用MLH1dn、PEmax和优化的epegRNA,显著提高了PE系统的编辑效率。此外,添加沉默突变到目标序列中可以消除DNA错配,从而提高靶向编辑的效率(见图9F)。
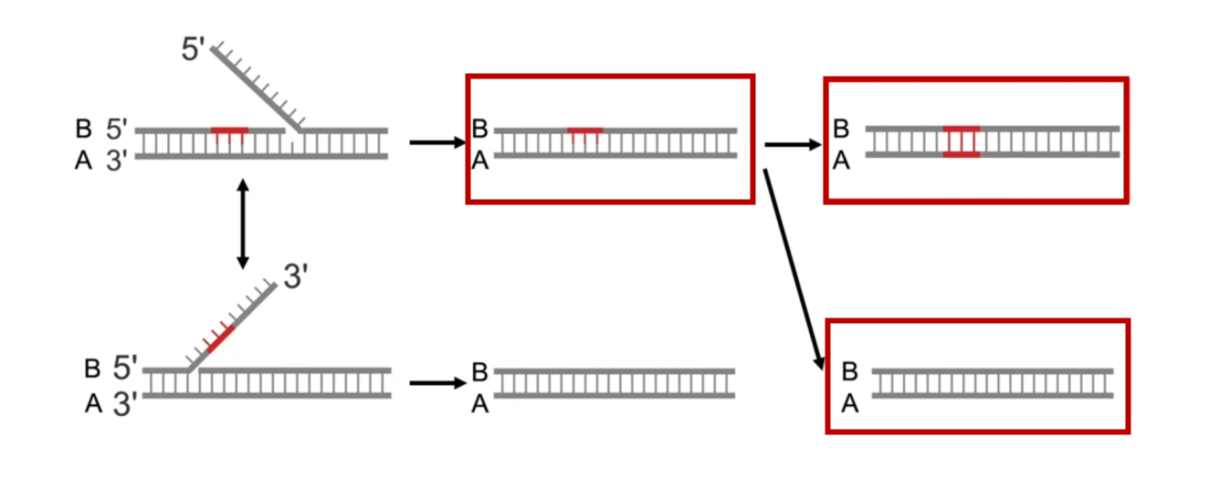
图10
本文主要介绍PE相关应用及近期提高该编辑系统效率的研究进展。通过优化pegRNA和PE效应蛋白两个方面来提高编辑效率,同时采用协同优化策略。尽管该技术仍处于早期发展阶段,但未来有望成为基因编辑领域的重要工具。随着技术的不断改进,PE技术将在育种、疾病治疗等领域取得重大突破。而秀丽线虫作为一种经典的模式生物,有望通过PE编辑来精确修正秀丽线虫中与疾病相关或影响生长发育的基因变异,或者快速生成特定基因型的药物测试模型等。目前我司也在着手进行PE编辑应用于线虫的相关研究,并且初步取得了一些成果。此外,我们还探索了不同的编辑系统组合,以期进一步提高编辑的精准性和稳定性。未来,我们将继续深入研究PE技术在秀丽线虫中的应用,并力争在基因功能研究、药物筛选以及疾病模型构建等领域取得更广泛的应用成果。我们期待通过不断的技术创新和优化,推动PE技术在基因编辑领域的广泛应用。
最后小编将PE的详细优化策略放在附表中,以期为大家的实验提供一些参考。

附表
参考文献
[1]Anzalone, A. V., Randolph, P. B., Davis, J. R., Sousa, A. A., Koblan, L.W., Levy, J. M.,et al. (2019). Search-and-replace genome editing without double-strand breaks or donor DNA. Nature 576 (7785), 149–157. doi:10.1038/s41586-019-1711-4
[2]Hassan, M. M., Zhang, Y., Yuan, G., De, K., Chen, J. G., Muchero, W., et al. (2021).Construct design for CRISPR/Cas-based genome editing in plants. Trends Plant Sci. 26(11), 1133–1152. doi:10.1016/j.tplants.2021.06.015
[3] Tong, Y., Jørgensen, T. S., Whitford, C. M., Weber, T., and Lee, S. Y. (2021). Aversatile genetic engineering toolkit for E. coli based on CRISPR-prime editing. Nat. Commun. 12 (1), 5206. doi:10.1038/s41467-021-25541-3
[4]Zhuang, Y., Liu, J., Wu, H., Zhu, Q., Yan, Y., Meng, H., et al. (2022). Increasing the efficiency and precision of prime editing with guide RNA pairs. Nat. Chem. Biol. 18 (1),29–37. doi:10.1038/s41589-021-00889-1
[5]Tao, R.,Wang, Y., Jiao, Y., Hu, Y., Li, L., Jiang, L., et al. (2022b). Bi-PE: Bi-directional priming improves CRISPR/Cas9 prime editing in mammalian cells. Nucleic Acids Res.50 (11), 6423–6434. doi:10.1093/nar/gkac506
[6]Lin, Q., Jin, S., Zong, Y., Yu, H., Zhu, Z., Liu, G., et al. (2021). High-efficiency prime editing with optimized, paired pegRNAs in plants. Nat. Biotechnol. 39 (8), 923–927.doi:10.1038/s41587-021-00868-w
撰稿:黄河
审核:陈岚彬
编辑:余雯