
点击蓝字 关注我们
对模式生物的研究揭示了自然遗传差异如何塑造性状变异,而这些发现得益于基因组可用性的不断增长以及研究人员对实验工具集的不断拓展。近年来,秀丽隐杆线虫越来越多地被用于利用定量遗传学来识别和测量自然遗传变异对性状的影响。自2016年以来,秀丽线虫自然多样性资源(C. elegans Natural Diversity Resource,CeNDR)通过提供野生菌株档案、每个菌株的全基因组序列和变异数据以及全基因组关联分析(GWA)图谱,促进了大约150项自然变异研究,包括全球种群结构的调查[1]、基因组内高度分化区域的进化起源[2]以及使用定量遗传图谱发现影响生态和生物医学相关性状的变异[3]。
原开发团队在CeNDR平台的基础上推陈出新,更新了最新的资源库即隐杆线虫属自然多样性资源(Caenorhabditis Natural Diversity Resource,CaeNDR),该平台能够对三种隐杆线虫属物种进行定量遗传学和基因组学研究,它们分别是:C. elegans、C. briggsae和C. tropicalis。CaeNDR 平台拥有多个数据库,这些数据库通过添加新菌株、全基因组序列数据和带注释的突变体而不断更新。此外,CaeNDR 还提供了新的交互式工具来探索自然变异并实现GWA映射。
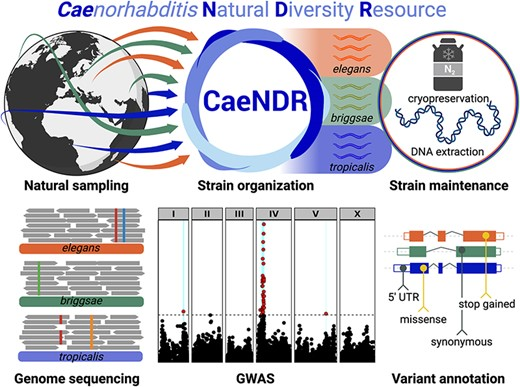
针对线虫的比较研究是目前非常热门的课题之一,其中包括自我受精的进化,参与外阴发育的细胞命运的调节,生殖细胞系发育的热耐受性和热可塑性,精子大小的发育决定因素,dauer形成的调节,信息素信号和信息素的生物合成等等。然而这些比较研究的许多见解仅基于每个物种内的少数菌株,甚至单个菌株。最常用的菌株是C. elegans的N2、C. briggsae的AF16和C. tropicalis的JU1373。而新的CaeNDR平台提供了更全面的隐杆线虫属菌株数据以方便科研人员的使用。
CaeNDR的三大功能:
1.
野生菌株的集中储存库
CaeNDR为隐杆线虫属研究提供了一个野生菌株的综合收集平台,研究人员或者公民科学家都可以通过网站提供的表单/链接向CaeNDR提供野生菌株。平台的目的是保存与野生菌株相关的生态数据,因此需要贡献者提供每个菌株的提交时间、地点、照片以及自然景观和基质的描述。
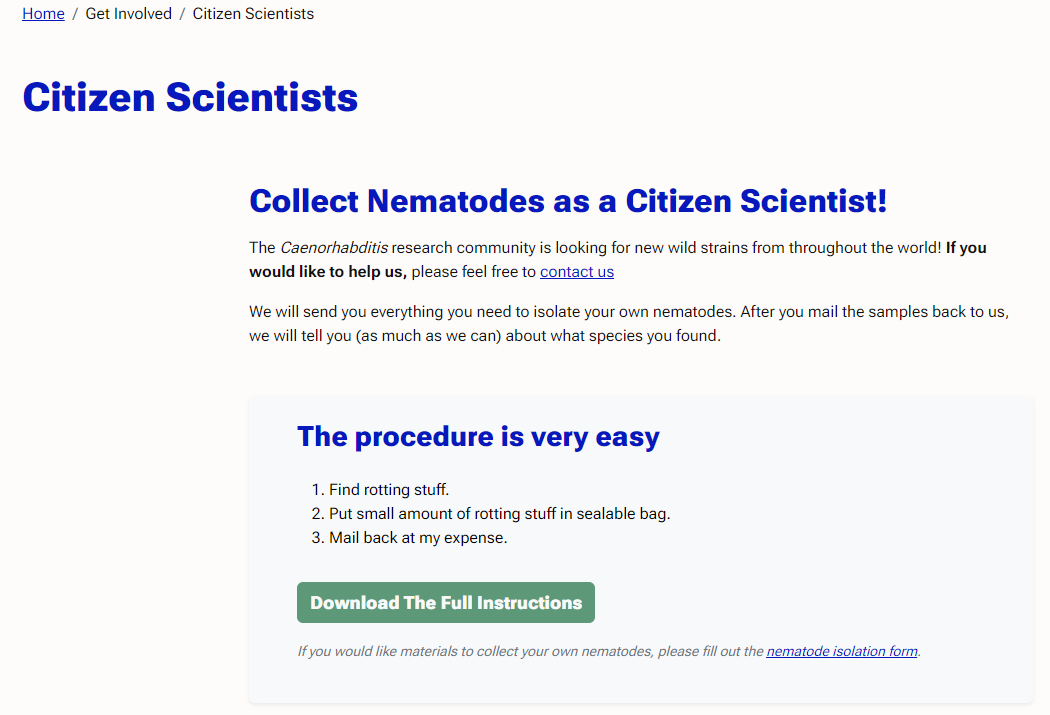
当然,研究人员也可以向CaeNDR单独或批量采购野生菌株,甚至包括平台预先选择的菌株集,以促进每个物种的GWAS研究。
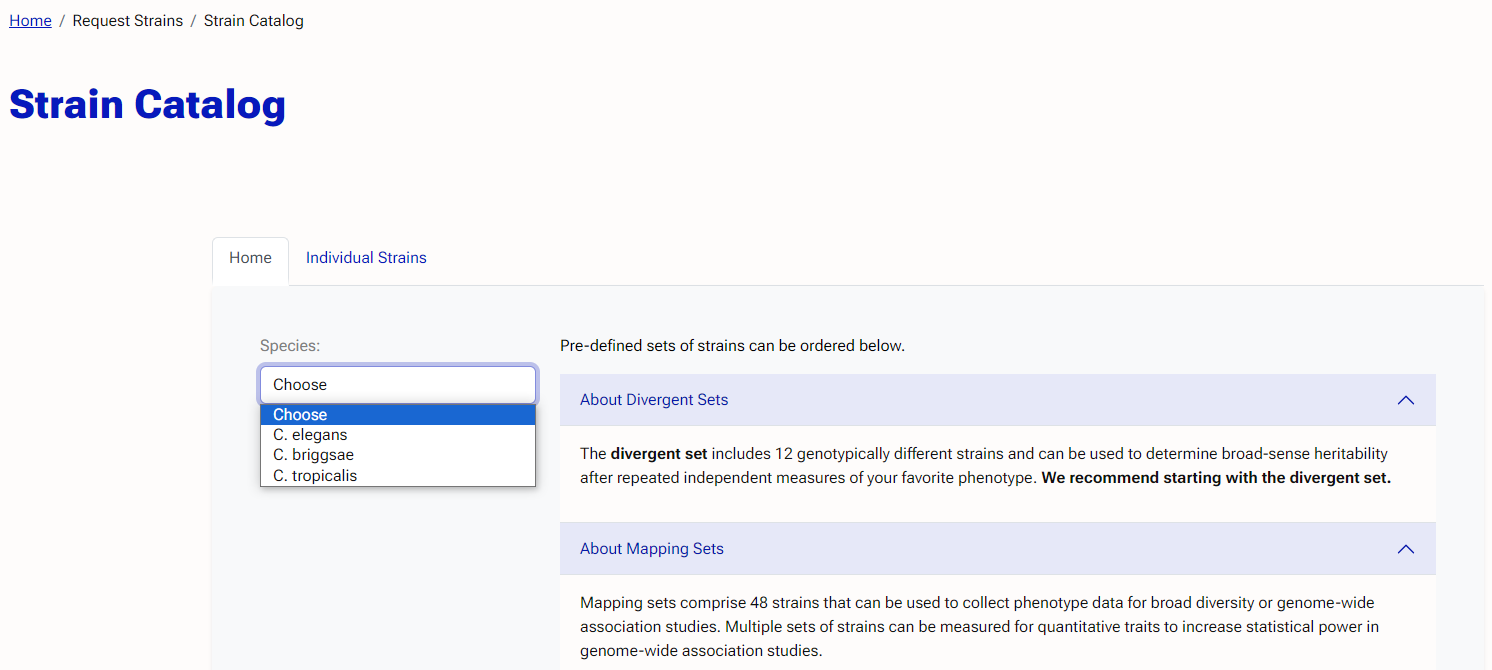
2.
隐杆线虫属的全物种基因组资源
CaeNDR维护并定期更新档案中菌株的全基因组序列和变异株数据。由于自交隐杆线虫主要在野外通过自我受精繁殖,因此从同一基质分离出的线虫通常几乎相同,只有少数差异可归因于近期的谱系特异性突变。包括GWAS在内的大多数CaeNDR工具都专注于探索同型之间的遗传变异。用于生成所有基因组数据集的软件和方法可在数据发布页面(caendr.org/data/data-release)上找到。
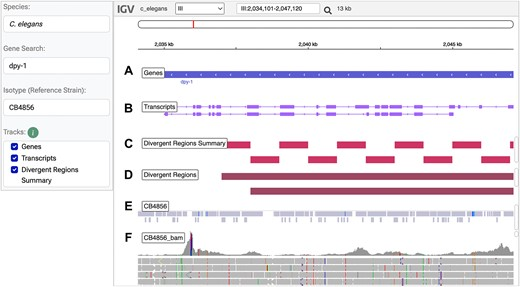
同时CaeNDR也提供一个类似wormbase的基因组浏览器工具,使用户能够可视化隐杆线虫属这三个物种中任何一个的基因组数据集。如下图所示,用户在左边的设置面板中指定物种。可选的“基因搜索”字段将引导浏览器指向基因组中的对应基因(A),并在下面的轨迹中显示转录本(B)。“分歧区域摘要”轨迹显示基因组中相对于该物种内任何同型参考菌株的参考基因组被归类为高分歧的区域(C),可以单击每个轨道特征来显示该特征所属的同种型参考菌株(D)。同型参考菌株的变异用蓝色(E)表示。BAM轨迹显示了被称为变异的读取水平数据。轨道的顶部显示读取深度,底部显示配对端读取(F)。
CaeNDR还能够支持研究人员通过突变体注释工具(caendr.org/tools/variant-annotation)来进一步研究某一特定区域,该工具显示了编码序列中的突变体如何在群体中分布,并提供了突变体如何影响蛋白质功能的预测。
3.
隐杆线虫属的GWAS工具箱
CaeNDR拥有多个基于网络的工具来帮助研究人员设计、执行和分析GWAS。一个典型的GWAS开始于一个试点研究,在独立重复的少量不同菌株中测量一个感兴趣的性状。然后,研究人员计算这些菌株之间由遗传差异解释的性状变异的比例,这被称为广义遗传度(H2)。通过使用遗传度计算器工具(caendr.org/tools/heritability-calculator),用户可以通过以Tab-Separated Values (TSV)文件的形式提交实验性实验的数据来快速估算H2。在GWAS菌株集中测量感兴趣的性状后,可以在CaeNDR的GWAS映射集中进行映射。基因图谱工具(caendr.org/tools/genetic-mapping)允许用户通过上传同型参考株性状测量的TSV文件来执行GWAS。
CaeNDR可以很好地被应用于自然变异的功能研究、群体基因组学和适应性变异研究及识别基因型-表型相关性研究等多个方面,是较为实用的线虫遗传学研究工具。平台目前仍在不断更新中,如果您对线虫遗传学研究感兴趣,不妨访问一下官网caendr.org/,了解更多关于CaeNDR的详细信息。
参考文献:
[1] Crombie T A, Zdraljevic S, Cook D E, et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations[J]. Elife, 2019, 8: e50465.
[2] Lee D, Zdraljevic S, Stevens L, et al. Balancing selection maintains hyper-divergent haplotypes in Caenorhabditis elegans[J]. Nature ecology & evolution, 2021, 5(6): 794-807.
[3] Burga A, Ben-David E, Lemus Vergara T, et al. Fast genetic mapping of complex traits in C. elegans using millions of individuals in bulk[J]. Nature communications, 2019, 10(1): 2680.
撰稿:陈岚彬
审核:林可敬
编辑:余雯

